Chapitre 5 Rcpp ou comment intégrer facilement du code C++dans un package R
Rcpp (R-C-Plus-Plus) est un package qui facilite l’interface entre C++ et R. R est un langage
interprété, ce qui facilite un certain nombre de choses (notamment nous donne accès à la console
dans laquelle on peut évaluer du code à la volée). Néanmoins, cette facilité d’utilisation se
compense entre autre par des temps de calcul supérieurs à ceux de langages de plus bas niveau,
tels que C, Fortran et C++ (mais qui nécessitent eux une compilation).
On dirigera le lecteur curieux vers le livre en ligne Rcpp for everyone de Masaki E. Tsuda, qui constitue une ressource très complète pour comprendre l’utilisation de Rcpp en plus de l’introduction que l’on peut trouver dans le livre Advanced R d’Hadley Wickham.
5.1 Première fonction en Rcpp
A vous de jouer !
- Afin de rendre votre package prêt pour l’utilisation avec Rcpp, commencez par executer la commande suivante :
devtools::use_rcpp()
Constatez les changements apportés
il faut également ajouter les 2 commentaires roxygen suivants dans la page d’aide du package dans son ensemble :
#' @useDynLib mypkgr
#' @importFrom Rcpp sourceCpp, .registration = TRUE
NULLNous allons maintenant créer une première fonction en Rcpp permettant d’inverser une matrice.
Pour cela, nous allons nous appuyer sur la library C++ Armadillo.
Il s’agit d’une library d’algèbre linéaire moderne et simple, hautement optimisée, et interfacée
avec R via le package RcppArmadillo.
C++ n’est pas un langage très différent de R. Les principales différences qui nous concernent :
C++est très efficaces pour le boucles for (y compris les boucles for emboîtées). Attention : il y a souvent un sens qui est plus rapide que l’autre (ceci est dû à la manière dont C++ attribue et parcours la mémoire).Chaque commande doit se terminer par un point virgule ‘;’
C++est un langage typé : il faut déclarer le type de chaque variable avant de pouvoir l’utiliser.
A vous de jouer !
Créez un nouveau fichier
C++depuis RStudio (via le menuFile>New File>C++ File), et enregistrez le dans le dossiersrc. Prenez le temps de le lire et essayez de comprendre chaque ligne.Compilez et chargez votre package (via le bouton “Install and Restart”) et essayez d’utiliser la fonction
timesTwo()depuis la console.Installez le package RcppArmadillo, et n’oubliez pas de faire les ajouts nécessaires dans
DESCRIPTION(cf.usethis::use_rcpp_armadillo())À l’aide de l’[introduction à
Rcpp]](http://adv-r.had.co.nz/Rcpp.html#rcpp-intro) de Hadley Wickham dans son livre Advanced R, ainsi que de la documentation du packageRcppArmadillode celle de la libraryC++Armadillo, tentez d’écrire une courte fonctioninvCenC++calculant l’inverse d’une matrice.Lorsque vous avez réussi à compiler votre fonction
invCet qu’elle est accessible depuis créer une fonctionmvnpdf_invC()à partir de l’implémentation demvnpdfsmarten remplaçant uniquement les calculs d’inverse matriciel par un appel àinvC.Evaluer le gain en performance de cette nouvelle implémentation
mvnpdf_invC.
n <- 1000
mb <- microbenchmark(mvtnorm::dmvnorm(matrix(1.96, nrow = n, ncol = 2)),
mvnpdf(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
mvnpdfsmart(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
mvnpdfoptim(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
mvnpdf_invC(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
times=100L)
mb## Unit: microseconds
## expr min
## mvtnorm::dmvnorm(matrix(1.96, nrow = n, ncol = 2)) 44.977
## mvnpdf(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE) 3315.752
## mvnpdfsmart(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE) 2317.976
## mvnpdfoptim(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE) 1749.224
## mvnpdf_invC(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE) 2318.017
## lq mean median uq max neval cld
## 67.3425 80.78763 79.8065 94.7715 136.284 100 a
## 3452.2000 3814.73348 3520.5470 3737.4780 11503.985 100 b
## 2375.6835 2546.84825 2398.5820 2443.0670 6170.336 100 c
## 1812.3845 1903.67961 1851.9495 1905.3520 5399.454 100 d
## 2361.3335 2510.35825 2390.5665 2434.7645 6297.723 100 c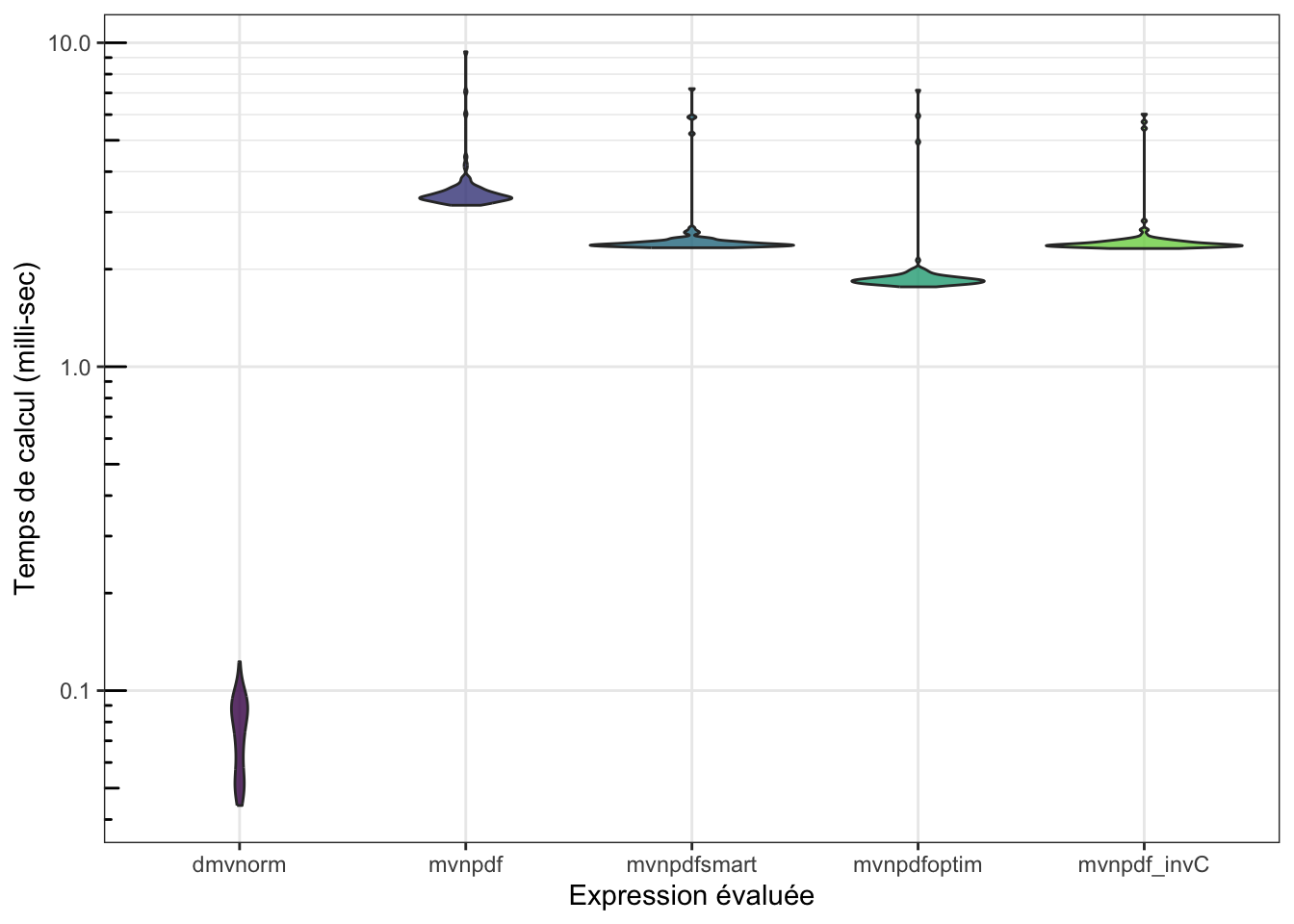
5.2 Optimisation grâce à C++
En règle générale, on ne gagne pas beaucoup en temps de calcul en remplaçant une fonction R
optimisée par une fonction en C++. En effet, la plupart des fonctions de base de R s’appuie en
réalité déjà sur des routines C ou Fortran bien optimisée. Le gain se limite alors simplement
à la suppression des vérifications des arguments et de la gestion des différents types.
A vous de jouer !
À partir de
mvnpdfsmart, proposez une implémentation completement enC++du calcul de densité de la loi Normale multivariéemvnpdfC().Evaluer le gain en performance de cette nouvelle implémentation
mvnpdfC
Vous pouvez télécharger notre proposition de mvnpdfC.cpp ici.
n <- 1000
mb <- microbenchmark(mvtnorm::dmvnorm(matrix(1.96, nrow = n, ncol = 2)),
mvnpdf(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
mvnpdfsmart(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
mvnpdfoptim(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
mvnpdf_invC(x=matrix(1.96, nrow = 2, ncol = n), Log=FALSE),
mvnpdfC(x=matrix(1.96, nrow = 2, ncol = n), mean = rep(0, 2), varcovM = diag(2), Log=FALSE),
times=100L)
mb## Unit: microseconds
## expr
## mvtnorm::dmvnorm(matrix(1.96, nrow = n, ncol = 2))
## mvnpdf(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE)
## mvnpdfsmart(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE)
## mvnpdfoptim(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE)
## mvnpdf_invC(x = matrix(1.96, nrow = 2, ncol = n), Log = FALSE)
## mvnpdfC(x = matrix(1.96, nrow = 2, ncol = n), mean = rep(0, 2), varcovM = diag(2), Log = FALSE)
## min lq mean median uq max neval cld
## 44.608 57.5025 78.25465 80.1140 93.7260 123.287 100 a
## 3340.475 3477.2920 3745.40330 3543.2405 3661.5665 8599.627 100 b
## 2322.199 2375.3350 2482.04570 2407.9505 2439.4385 6698.129 100 c
## 1745.903 1823.2085 2027.96824 1863.0400 1915.9505 6711.659 100 d
## 2317.074 2366.0895 2516.76532 2402.1285 2439.4385 6947.655 100 c
## 35.506 43.5625 55.14500 47.0475 53.5255 713.441 100 a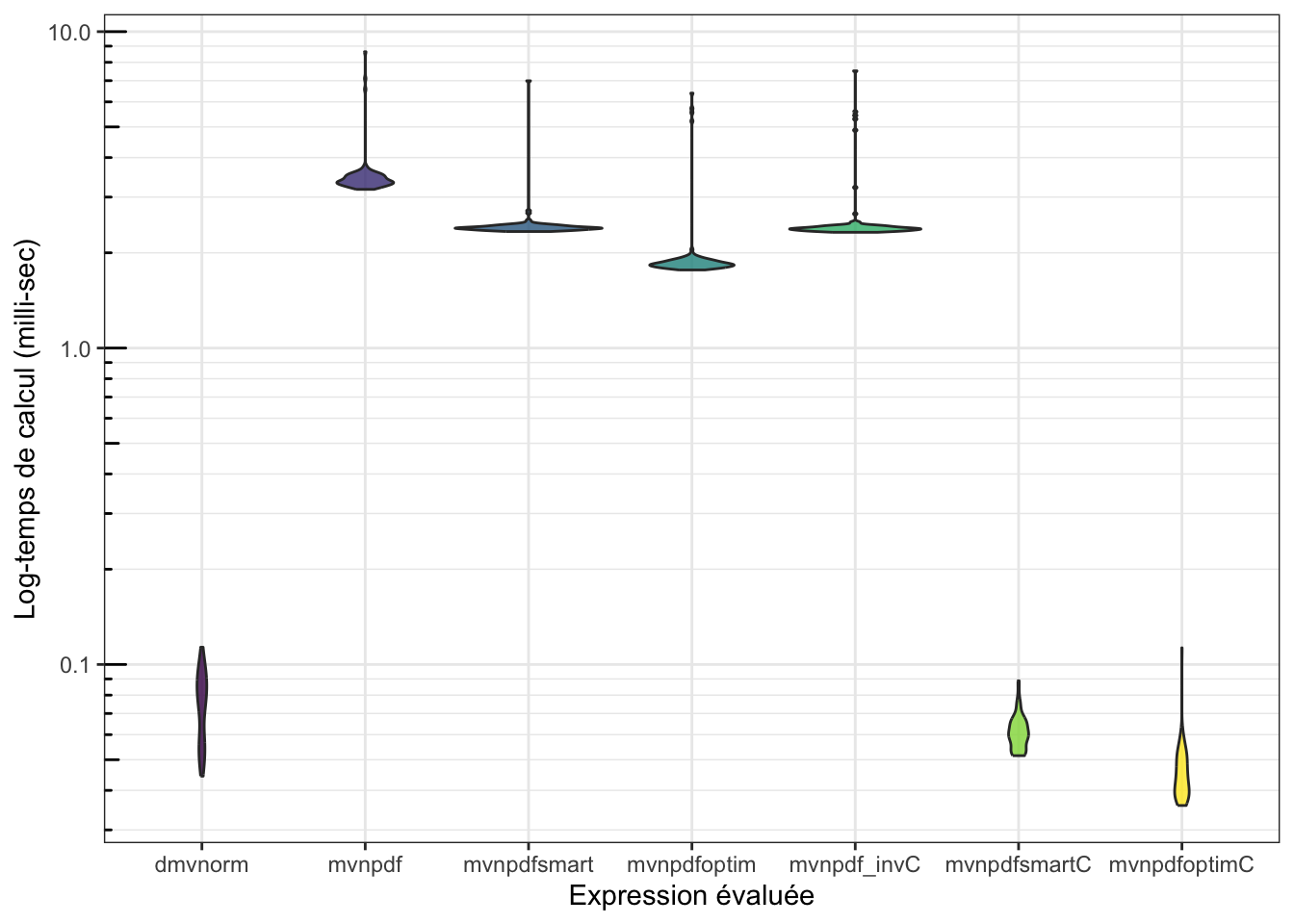
À noter que vous pouvez utiliser des fonctions Rcpp en dehors de l’architecture
d’un package grâce à la fonction Rcpp::sourceCpp(). Mais comme nous avons
qu’il est préférable de gérer tous ces code sous la forme de package, il est
peu probable que vous en ayez besoin !
5.3 Annexe 5.1 : l’optimisation prématurée n’est pas une bonne idée
Chambers, Software for Data Analysis: Programming with R, Springer, 2008 :
Including additional C code is a serious step, with some added dangers and often a substantial amount of programming and debugging required. You should have a good reason.